6. 비지도 학습
6.1. 군집 알고리즘(Clustering)
6.1.1. 개요 - 비지도학습이란

- 비지도 학습
- 사용자가 기계에 특성 데이터만 알려주고, 정답을 가르쳐주지 않는 학습 알고리즘
- 즉, 정답을 알려주지 않아도 기계가 데이터에 있는 구조나 패턴을 스스로 찾아내는 알고리즘
- 대표적으로 군집(Clustering)과 차원 축소(Dimension Reduction) 방법이 있음
- 군집(clustering) : 비슷한 샘플끼리 그룹(cluster)으로 모으는 작업
- 차원 축소 (Dimension Reduction) : 특성 개수를 줄이는 작업
6.1.2. 과일 분류하기 코드
- 과일 사진 데이터 로드 및 확인
import numpy as np
import matplotlib.pyplot as plt
#가로x세로 100x100 크기의 300개의 이미지가 담긴 npy 배열 로드
fruits = np.load('fruits_300.npy')
print(fruits.shape) # 결과 : (300, 100, 100)
#fruits의 값을 출력해보면 물체가 있는 부분이 255에 가깝고, 물체가 없는 부분(배경)은 0에 가까움
#컴퓨터는 물체에 집중을 해야하는데, 물체가 있는 부분이 0이면 계산이 어렵기 때문
print(fruits[0, 0, :])
#imshow => image show. numpy 배열을 이미지로 보여주는 메소드
#cmap => 이미지로 보여줄 때 사용할 컬러의 종류를 지정할 수 있음. 기본값은 밝은 녹색
plt.imshow(fruits[0], cmap='gray')
plt.show()
plt.imshow(fruits[0], cmap='gray_r')
plt.show()
#subplots => 한 번에 여러개의 그래프를 확인하고 싶을 때 사용하는 메소드
fig, axs = plt.subplots(1, 2)
axs[0].imshow(fruits[100], cmap='gray_r')
axs[1].imshow(fruits[200], cmap='gray_r')
plt.show()

- 이미지 픽셀 값 분석하기
#reshape 메소드를 통해 2차원 배열을 1차원으로, 즉 일렬로 나란히 만듦
apple = fruits[0:100].reshape(-1, 100*100)
pineapple = fruits[100:200].reshape(-1, 100*100)
banana = fruits[200:300].reshape(-1, 100*100)
print(apple.shape) #결과 : (100,10000)
#히스토그램으로 픽셀을 차지하는 평균값 확인
#legend => 범례를 만들어주는 메소드
#axis => 2차원 배열일 때 0일때는 행 방향(세로) 계산, 1일때는 열 방향(가로)계산
plt.hist(np.mean(apple, axis=1), alpha=0.8)
plt.hist(np.mean(pineapple, axis=1), alpha=0.8)
plt.hist(np.mean(banana, axis=1), alpha=0.8)
plt.legend(['apple', 'pineapple', 'banana'])
plt.show()
#막대 그래프로 각 픽셀의 평균값 확인
fig, axs = plt.subplots(1, 3, figsize=(20, 5))
axs[0].bar(range(10000), np.mean(apple, axis=0))
axs[1].bar(range(10000), np.mean(pineapple, axis=0))
axs[2].bar(range(10000), np.mean(banana, axis=0))
plt.show()

- 픽셀 평균값으로 이미지 그리기
#이미지를 그리기 위해 1차원 배열을 다시 2차원 배열로 변경
apple_mean = np.mean(apple, axis=0).reshape(100, 100)
pineapple_mean = np.mean(pineapple, axis=0).reshape(100, 100)
banana_mean = np.mean(banana, axis=0).reshape(100, 100)
fig, axs = plt.subplots(1, 3, figsize=(20, 5))
axs[0].imshow(apple_mean, cmap='gray_r')
axs[1].imshow(pineapple_mean, cmap='gray_r')
axs[2].imshow(banana_mean, cmap='gray_r')
plt.show()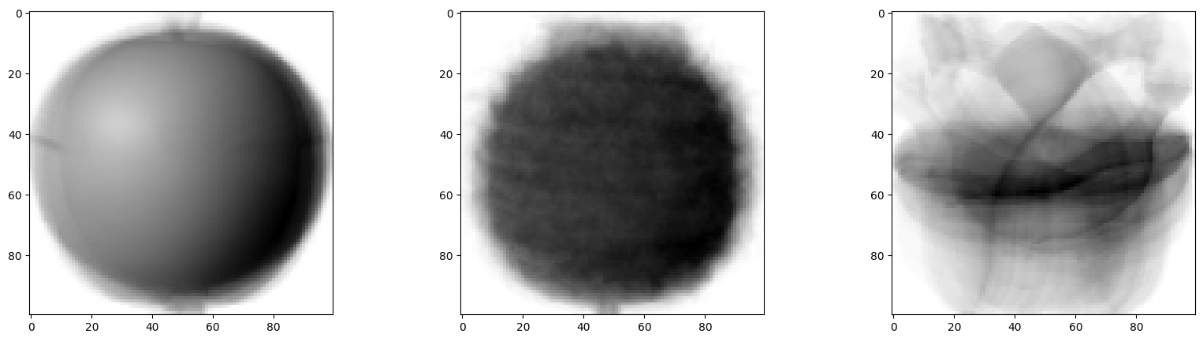
- 평균값과 가까운 이미지 고르기
#절댓값 오차를 통해 평균값과의 차이 계산
abs_diff = np.abs(fruits - apple_mean)
abs_mean = np.mean(abs_diff, axis=(1,2))
print(abs_mean.shape)
#argsort => 작은 값에서 큰 값 순서대로 정렬후 반환하는 메소드 => 차이가 작을수록 더 가까운(비슷한) 것
#axis('off') 옵션 => 그래프 그릴 시 축을 나타내지 않는 옵션
apple_index = np.argsort(abs_mean)[:100]
fig, axs = plt.subplots(10, 10, figsize=(10,10))
for i in range(10):
for j in range(10):
axs[i, j].imshow(fruits[apple_index[i*10 + j]], cmap='gray_r')
axs[i, j].axis('off')
plt.show()
6.2. K-평균
6.2.1 개요 - K-평균 알고리즘 (K-means)
- 앞선 예제에서는 각 사진이 어떤 과일인지 알고 있어서 각 과일의 평균값을 직접 구할 수 있었으나 실전은 타겟을 모름 => K-means 알고리즘을 사용해서 해결
- K-means 알고리즘
- 평균값, 즉 클러스터의 중심(센트로이드(centroid))을 찾아주는 알고리즘
- k ⇒ 몇 개의 군집을 찾을것인지 (k개) 정하는 k-means의 하이퍼파라미터
- k-means는 원형의 데이터들에 적합한 알고리즘임 (이에 동그랗게 데이터 전처리를 하고 사용하면 좋음)
- K-means 작동 방식

- 무작위로 k개의 클러스터 중심을 찍음
- 각 샘플에서 가장 가까운 클러스터 중심을 찾아 해당 클러스터의 샘플로 지정
- 클러스터에 속한 샘플의 평균값으로 클러스터 중심을 변경
- 클러스터 중심의 위치가 많이 안 변할 때까지 2번으로 돌아가 반복
6.2.2 K-means 코드
#2차원 배열로 데이터 준비
import numpy as np
fruits = np.load('fruits_300.npy')
fruits_2d = fruits.reshape(-1, 100*100)
#n_clusters => 몇 개의 클러스터를 만들지 설정
#비지도 학습임에 fit에 타겟 데이터를 넣지 않음
#kMeans의 결과는 labels_에 저장되어 있음
from sklearn.cluster import KMeans
km = KMeans(n_clusters=3, random_state=42)
km.fit(fruits_2d)
print(km.labels_) # 결과 : [0, 0, 2, 1, 2 (생략)] / 레이블 값과 순서에는 아무런 의미 X, 그저 비슷한 걸 묶은 것
#군집당 몇개씩 나누어졌는지 출력
print(np.unique(km.labels_, return_counts=True)) #결과 : (array([0, 1, 2]), array([111, 98, 91], dtype=int64))
#분류가 제대로 됐는지 분류된 이미지들을 출력해서 확인하는 코드
#실전에서는 데이터가 크기때문에 클러스터가 어떻게 나뉘었는지 전부 보기 어려움 => 샘플링 같은 기법 사용
import matplotlib.pyplot as plt
def draw_fruits(arr, ratio=1):
n = len(arr)
rows = int(np.ceil(n/10))
cols = n if rows < 2 else 10
fig, axs = plt.subplots(rows, cols,
figsize=(cols*ratio, rows*ratio), squeeze=False)
for i in range(rows):
for j in range(cols):
if i*10 + j < n:
axs[i, j].imshow(arr[i*10 + j], cmap='gray_r')
axs[i, j].axis('off')
plt.show()- 클러스터 중심 찾기
#km.cluster_centers_ => 센트로이드의 데이터값이 저장되어있는 메소드
draw_fruits(km.cluster_centers_.reshape(-1, 100, 100), ratio=3)
- 샘플별 클러스터 중심까지의 거리 구하기
#transform 메소드 => 훈련 데이터 샘플에서 클러스터 중심까지 거리를 변환해 줌. 이를 특성으로 사용할 수 있음
#이를 활용하면 지도 학습때 데이터셋의 차원을 줄일 수도 있음
#predict 메소드 => 선택한 샘플의 가장 가까운 클러스터 중심을 기준으로 클래스를 예측하는 메소드
print(km.transform(fruits_2d[100:101])) #결과 : [[3393.8136117 8837.37750892 5267.70439881]]
print(km.predict(fruits_2d[100:101])) #결과 : [0]6.2.3 최적의 K(클러스터 개수) 찾기
- 이너셔(inertia)
- 클러스터 중심과 클러스터에 속한 샘플 사이의 거리를 제곱 합한 값
- 즉, 클러스터에 속한 샘플들이 얼마나 밀집되어 있는지를 나타내는 값이라 볼 수 있음.
- 엘보우 방법(elbow)
- k를 작은값에서 큰 값으로 바꿔가며 이너셔의 변화를 관찰하여 최적의 k를 찾는 방법
- 이너셔 값이 꺾어지는 지점(팔꿈치 처럼 생긴 지점)의 값이 비교적 좋은 k임
inertia = []
for k in range(2, 7):
km = KMeans(n_clusters=k, n_init=10, random_state=42)
km.fit(fruits_2d)
inertia.append(km.inertia_)
plt.plot(range(2, 7), inertia)
plt.xlabel('k')
plt.ylabel('inertia')
plt.show()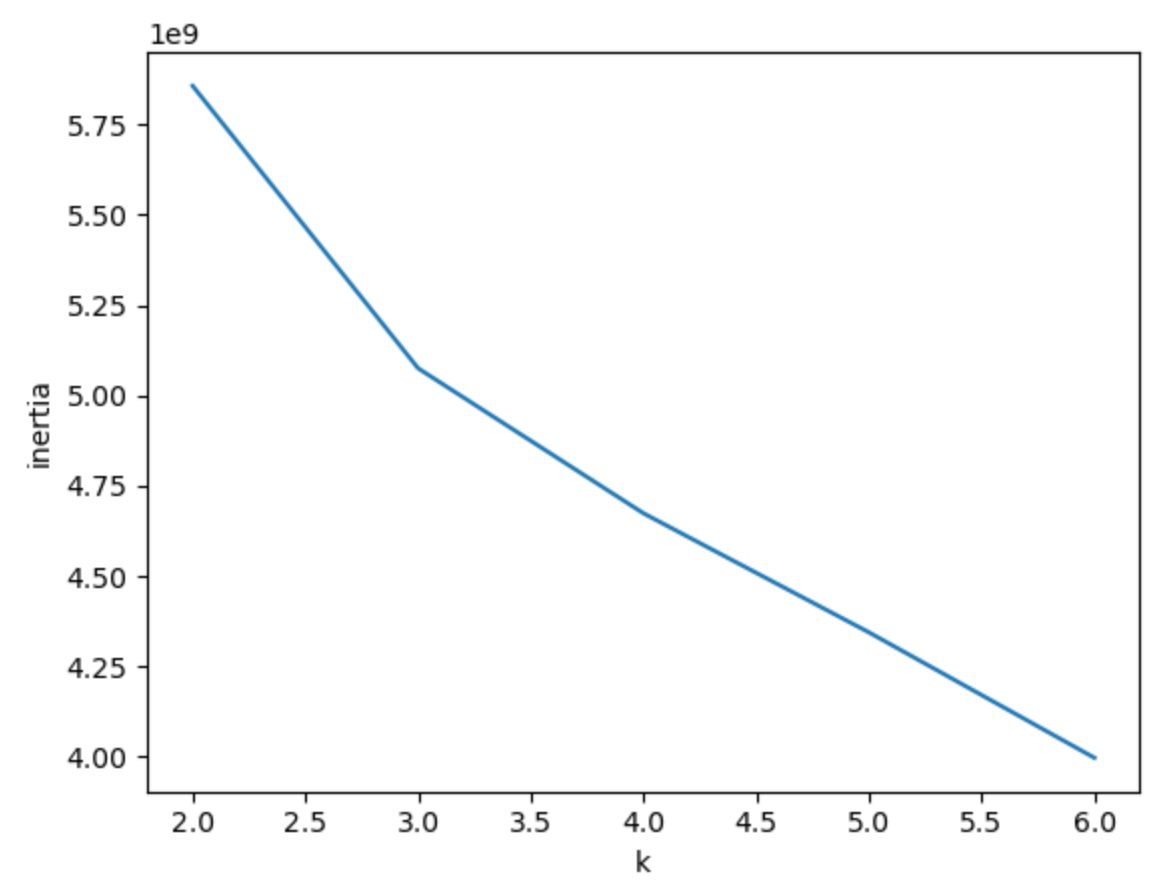
6.3. 주성분 분석
6.3.1. 개요 - 차원 축소 (Dimension reduction)
- 차원(Dimension)

- 1차원 배열(벡터)에서의 차원 ⇒ 원소의 개수
- 다차원 배열에서의 차원 ⇒ 축(특성)의 개수
- ex) 2차원 배열에서는 행과 열이 차원을 의미, 100x100의 과일 이미지는 10000개의 특성(10000개의 차원)
- 차원 축소(Dimension reduction)
- 원본 데이터의 특성을 적은 수의 새로운 특성으로 변환하는 비지도 학습 방법
- 차원 축소를 진행하면 저장 공간을 줄이고 시각화하기 쉬워짐. 차원 축소를 선행한 데이터를 다른 알고리즘에 사용하면 성능도 향상시킬 수 있음
6.3.2 주성분 분석 (PCA)
- 주성분 분석 (PCA, Principal Component Analysis)
- 대표적인 차원 축소 알고리즘
- 데이터에서 가장 분산이 큰 방향(주성분)을 찾는 알고리즘
- 원본 데이터를 주성분에 투영해 새로운 특성을 만들 수 있음
- 주성분의 정의 및 특징 예시
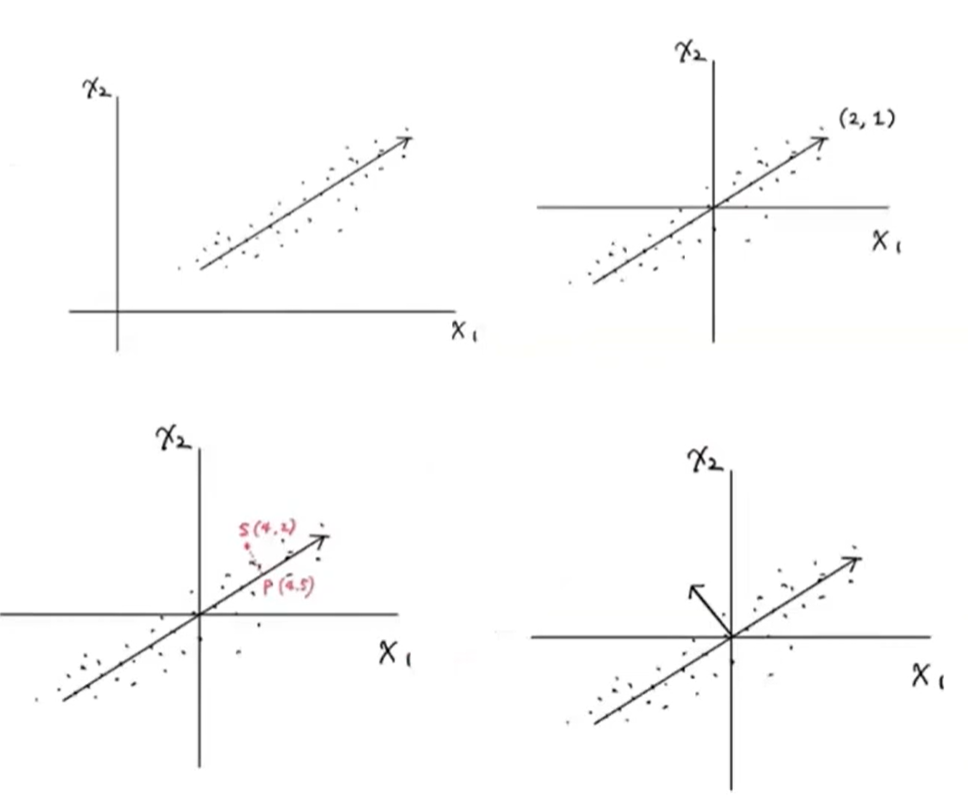
- 그림 (1) - 그림에 나타난 대각선이 현재 2차원 데이터를 가장 잘 표현하는 대각선
- 그림 (2) - 해당 대각선이 원점에서 출발한다면 두 원소로 이루어진 벡터 (2,1) 처럼 표현할 수 있음
- 해당 벡터를 주성분(원본 데이터에 있는 어떤 방향)이라고 함
- 주성분 벡터의 개수는 원본 데이터셋에 있는 특성 개수와 같음
- 그림 (3) - 원본 데이터를 주성분의 수직인 방향으로 투영하면 차원을 축소할 수 있음
- 그림 (3) 에서는 s(4,2) 이라는 샘플 데이터를 넣어 p(4.5) 라는 1차원 데이터를 산출해냄
- 주성분이 가장 분산이 큰 방향이기 때문에 주성분에 투영한 데이터는 원본이 가지고 있는 특성을 가장 잘 나타냄
- 그림(4) - 하나의 주성분을 산출해낸 뒤 그 주성분에 수직이고 분산이 가장 큰 다음 방향을 찾으면 그 다음 주성분을 찾을 수 있음 (수직이 아닌 것은 비효율적임)
6.3.3 PCA 코드
- 주성분 찾기
#2차원 배열로 데이터 준비
import numpy as np
fruits = np.load('fruits_300.npy')
fruits_2d = fruits.reshape(-1, 100*100)
#PCA의 n_components => 우리가 찾을 주성분의 개수
#pca의 fit 메소드에도 비지도 학습이기 때문에 타겟값 제공하지 않음
from sklearn.decomposition import PCA
pca = PCA(n_components=50)
pca.fit(fruits_2d)
#components_ => PCA가 찾아낸 주성분을 저장해놓음
print(pca.components_.shape) #결과 : (50, 10000) / 50개의 주성분 중에 원소의 개수는 10000개- 주성분 기준 원본 데이터 변환
#PCA의 transform 메소드 => 입력한 주성분을 투영하여 원본 데이터의 차원을 줄임
fruits_pca = pca.transform(fruits_2d)
print(fruits_pca.shape) #결과 : (300, 50) / 10000개의 특성을 50개로 줄임- 원본 데이터 복원
#inverse_transform 메소드 => 압축했던 걸 약간의 손실을 감안하면 복원할 수 있음
fruits_inverse = pca.inverse_transform(fruits_pca)
print(fruits_inverse.shape) # 결과 : (300, 10000) / 특성이 10000으로 돌아옴
fruits_reconstruct = fruits_inverse.reshape(-1, 100, 100)
for start in [0, 100, 200]:
draw_fruits(fruits_reconstruct[start:start+100])
print("\\n")
- 설명된 분산(explained variance)
# 설명된 분산 => 각 주성분이 얼마나 원본 데이터의 분산을 잘 설명하는지 나타내는 값
# explained_variance_ratio_ => 각 주성분의 설명된 분산 비율을 기록해놓는 메소드
print(np.sum(pca.explained_variance_ratio_)) #결과 : 0.9215353828621149 / 원본의 92% 만큼을 표현하고 있음
#설명된 분산 그래프를 통해 적절한 주성분 개수를 확인할 수 있음
plt.plot(pca.explained_variance_ratio_)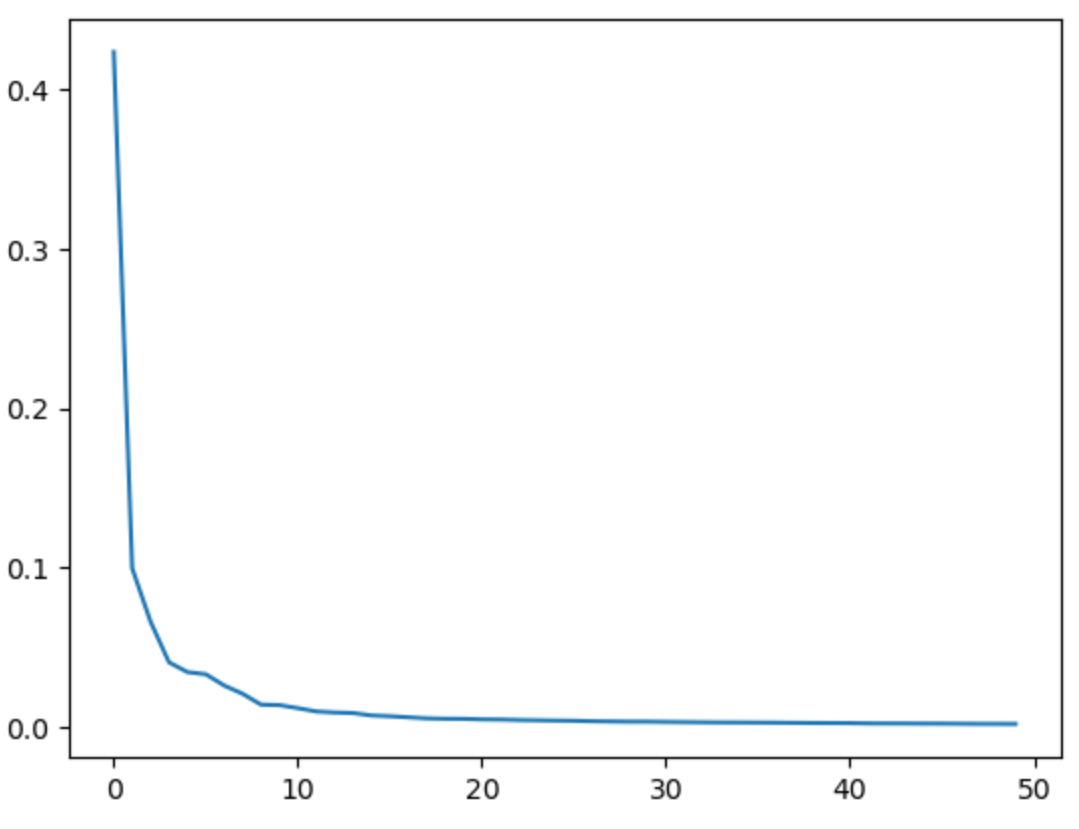
참고자료 :
1. 혼자 공부하는 머신러닝, 박해선, 한빛미디어
2. 혼자 공부하는 머신러닝 인프런 강의, 박해선, https://www.inflearn.com/course/%ED%98%BC%EC%9E%90%EA%B3%B5%EB%B6%80-%EB%A8%B8%EC%8B%A0%EB%9F%AC%EB%8B%9D-%EB%94%A5%EB%9F%AC%EB%8B%9D/dashboard
'Group Study (2024-2025) > Machine Learning 입문' 카테고리의 다른 글
| [ML 입문] 7주차 스터디 (1) | 2024.11.20 |
|---|---|
| [ML 입문] 6주차 스터디 (3) | 2024.11.10 |
| [ML 입문] 4주차 스터디 (0) | 2024.10.30 |
| [ML 입문] 3주차 스터디 (1) | 2024.10.16 |
| [ML 입문] 2주차 스터디 (0) | 2024.10.07 |